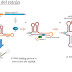
El pasado Lunes 17 de Enero, tuvimos una jornada “rápida” en la que ya vislumbramos la importancia biológica de lo que va a ser nuestra tarea: la descripción completa de un sistema de retrones no descrito hasta la fecha: el sistema III-A3.
Figura 1: Esquema del sistema de retrones tipo III-A3. |
Para ello, nos vamos a servir de 4 “subgrupos” de ejemplos (A-D) que tendremos que analizar “conjuntamente y por separado …
 |
| Figura 2: Grupos que vamos a estudiar según la filogenia de las RTs. Son grupos de genes relacionados entre sí de manera significativa (valor de relación muy cercano al 1). |
Y hemos realizado las comparaciones de los dos genes: el efector (PRTaseWH) y la propia reverso transcriptasa (RT). Observando algunas peculiaridades (Figura 3)
 |
| Figura 3: Comparativa de las 5 proteínas más relacionadas con nuestro locus modelo (1352 de Salmonella enterica). |
En realidad, se debe a un error en nuestra interpretación, la proteína (el gen) efectora del locus 155 tiene un tamaño semejante a las otras. Entonces, ¿por qué el clone manager nos la identifica más pequeña?
 |
| Figura 4: la herramienta “find ORFs” (búsqueda de marcos abiertos de lectura) del clone manager. |
Como podemos observar en el programa la búsqueda de marcos abiertos de lectura no comienza siempre con el triplete ATG… . De hecho, aunque es el mayoritario y más eficiente, no es el único.
Os recomiendo que le echemos un vistazo a este trabajo:
Hecht A, Glasgow J, Jaschke PR, Bawazer LA, Munson MS, Cochran JR, Endy D, Salit M. Measurements of translation initiation from all 64 codons in E. coli. Nucleic Acids Res. 2017 Apr 20;45(7):3615-3626. doi: 10.1093/nar/gkx070. PMID: 28334756; PMCID: PMC5397182.
Que nos lo hemos descargado en recursos (enlace).
En él miden y comparan la eficiencia de comienzo de la traducción de todos y cada uno de los tripletes de nucleótidos (que son 64) (Figura 5).
 |
| Figura 5: la traducción puede comenzar por tres tripletes: AUG, GUG y UUG, aunque destaca mayoritariamente AUG. |
Nuevos subgrupos nos quedan por analizar… esta semana debemos anotarlos todos y “ordenarlos” según su parecido a nuestro sistema modelo: el 1352 de Salmonella entérica.
Primero, los 9 representantes del grupo B (1325-1333), continuaremos por los del C y el D.
¡Confío en que los tengamos todos identificados para la próxima reunión!!!!
Hip, hip hurra
Aupa DNA team!!!!





No hay comentarios:
Publicar un comentario