El pasado 20 de Diciembre tuvimos nuestra última reunión del año (que no la última del proyecto… jejeje).
En ella, vuestros padres conocieron donde andáis ya metidas. Creo que será bueno que también nos sigan en nuestro blog, verdad???
Tras una breve introducción de nuestro centro de investigación y los distintos proyectos caos de este año, de una manera somera les expliqué hasta donde queríamos llegar…
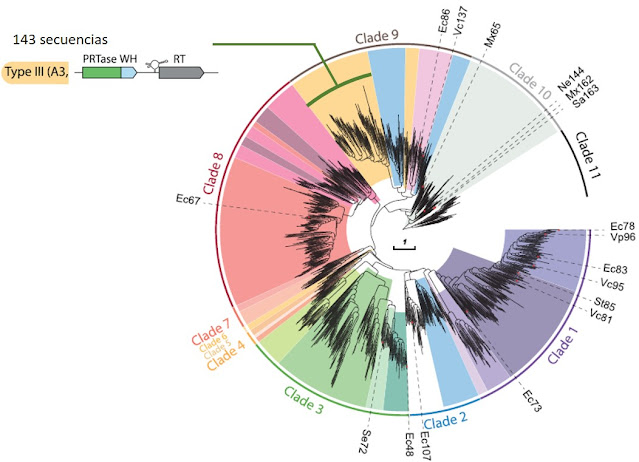
Figura resumen del artículo que nos servirá como modelo a seguir en el análisis de nuestras secuencias… .
Y es que el proyecto ya va cobrando forma. Es más ya sabemos que vamos a estudiar un grupo particular de retrones para los que todavía no hay una forma de RNA identificada (en recursos se ha subido la presentación correspondiente a este día). Y por tanto nuestro análisis permitirá validar su posible función biológica.
Figura 2: Arbol filogenético con todos los tipos de retrones que existen en la naturaleza. Se señalan las 143 secuencias sobre las que echaremos un ojo que representan a los retrones del tipo III-A3.
De momento tenemos como tarea el análisis de un subgrupo muy particular de retrones que hemos denominado como subgrupo A, correspondiente a 5 regiones del genoma de estas 5 bacterias (species strain en la tabla)
Y me consta que ya le habéis echado un ojo!!!!!